Rechenleistung spendenJeder PC-Nutzer kann Covid-19 bekämpfen
Wer nicht nur passiv durch Social Distancing im Kampf gegen das Coronavirus helfen möchte, kann sich mit Hunderttausenden anderen Computer-Nutzern zusammentun, um mit vereinten Rechen-Kräften die Covid-19-Forschung zu unterstützen. Es ist ganz einfach, jeder kann mitmachen.
Um mithilfe von Computermodellen und -simulationen Wirkstoffe und vielleicht schon bald einen Impfstoff gegen Covid-19 zu finden, ist jede Menge Rechenpower nötig. Deshalb haben sich bereits große US-Tech-Konzerne und Forschungseinrichtungen zum Covid-19-High-Performance Computing Consortium zusammengeschlossen, um ihre Supercomputer- und Cloud-Kapazitäten zu bündeln.
Gemeinsam sind wir die Stärksten
Aber auch jeder Einzelne kann seinen Beitrag leisten, indem er ungenutzte Rechenleistung seines Computers zur Verfügung stellt. Wie stark man gemeinsam sein kann, zeigt vor allem das Projekt Folding@home, dessen gebündelte Rechenpower mit inzwischen rund 475 Petaflops sogar die Kapazität der im Covid-19-High-Performance Computing Consortium vereinten 16 Supercomputer übertrifft
Folding@home wurde von der Stanford University bereits vor 20 Jahren ins Leben gerufen und gehört inzwischen zur Washington University in St. Louis. Das Projekt hat sich der Erforschung von Proteinen gewidmet. Genau genommen geht es dabei um deren dreidimensionale Strukturen und wie sie durch Faltungen zustande kommen.
Suche nach Mini-Protein-Bindern
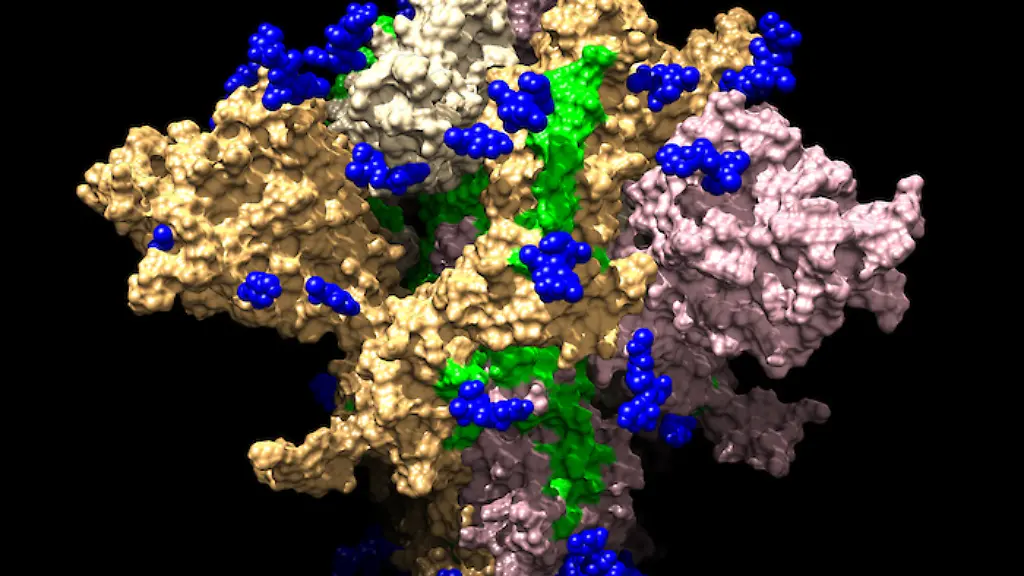
Proteine spielen bei der Infektion mit dem neuartigen Coronavirus (Sars-CoV-2) eine entscheidende Rolle. Es hat an der Oberfläche ein sogenanntes Spike-Protein, mit dem es an den Rezeptoren einer angegriffenen Zelle andockt. Die Erforschung dieses Prozesses könnte also von entscheidender Bedeutung sein.
Tatsächlich konnte ein Team um Folding@home-Direktor Greg Bowman bereits mithilfe der vereinten Nutzer-Rechenpower die Struktur des Sars-CoV-2-Spike-Proteins entschlüsseln, das aus drei einzelnen Proteinen zusammengesetzt ist. Jetzt geht es darum, Medikamente zu finden. Und auch hier meldet Folding@home auf Twitter erste Erfolge. Man habe Taschen im Spike-Protein entdeckt, an die sich Wirkstoffe binden könnten. Solche auch Mini-Protein-Binder genannten Moleküle würden verhindern, dass das Virus an Zellen andocken kann, und es so neutralisieren.
Um Teil der Folding-Gemeinschaft zu werden, genügt es, ein kleines Programm (Windows, Mac, Linux) zu installieren und zu starten. Die ungenutzten Rechen-Ressourcen zweigt es dann automatisch ab. Allerdings hat sich das Projekt in den vergangenen Tagen schon so stark herumgesprochen, dass Folding@home kaum hinterherkommt, seine Infrastruktur anzupassen. Es kann also unter Umständen ein Weilchen dauern kann, bis die Hilfe abgerufen wird.
BOINC und Rosetta@home
Folding@home ist aber nicht die einzige Möglichkeit, mit überschüssiger PC-Rechenleistung am Kampf gegen das Coronavirus teilzunehmen. Unter anderem kann man sich BOINC anschließen. Dabei handelt es sich um ein Projekt der kalifornischen Berkeley-Universität, das seit 2002 "Volunteer Computing" unter anderem zur Suche nach außerirdischem Leben (SETI) einsetzt. Rosetta@home setzt die BOINC-Rechenpower zur Protein-Forschung ein. Das Projekt wird vom Baker Lab der University of Washington in Seattle betrieben.
Spannend ist auch Foldit - Solve Puzzles for Science, das Nutzern die Möglichkeit gibt, sich spielerisch am Kampf gegen das Coronavirus zu beteiligen. Im Prinzip erpuzzelt man Proteinstrukturen, die gegen Sars-CoV-2 eingesetzt werden könnten. Man muss dafür keine medizinischen Vorkenntnisse haben. Die Forscher setzen bei den gestellten Aufgaben auf Lösungsideen, die ihnen mit ihrer akademischen Herangehensweise vielleicht entgangen wären. Die Resultate der Rätselspiele werden wiederum vom Institut für Protein-Design der University of Washington ausgewertet.